Modélisation du vivant
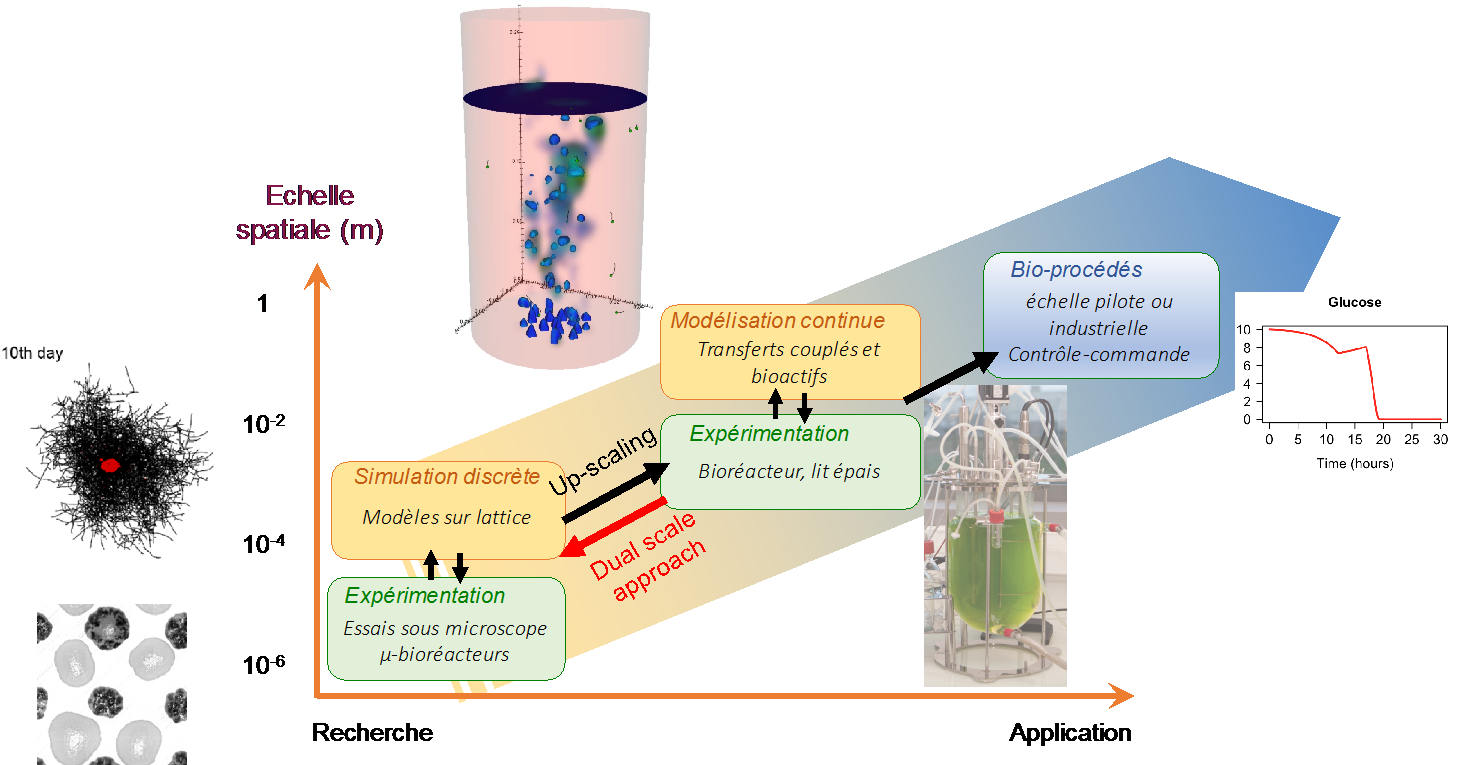
Dans le contexte LGPM, le périmètre du terme "modélisation du vivant" est limité aux bioprocédés, c'est-à-dire aux procédés faisant appel à des micro-organismes.
Ci-contre : positionnement du LGPM en modélisation du vivant.
Illustrations : cultures mixtes immobilisées de levures et µ-algues, modèle discret de développement de mycélium, modélisation CFD d'un photobioréacteur avec suivi lagrangien de µ-algues et champs spatiaux de concentrations, photobioréacteur de 5 litres et modélisation de croissance de levures.
Le LGPM développe deux stratégies complémentaires de modélisation. La première approche s'intéresse aux cultures en milieu liquide en supposant le bioréacteur parfaitement mélangé. Dans ce cas, la modélisation est 0D : les paramètres du modèle discret sont identifiés sur mesures globales et le modèle peut servir au contrôle-commande des bioréacteurs (1,2) ou pour comprendre le couplage entre populations de culture mixtes.
La seconde approche a pour ambition de dérouler une stratégie de changement d'échelle y compris sur le comportement du vivant. Cette trajectoire est plus longue à mettre en place car la complexité du modèle qui décrit le comportement du vivant doit être en adéquation avec la complexité de la modélisation du procédé. Par exemple, un photobioréacteur ne peut jamais être parfaitement mélangé car la lumière n'est pas homogène au sein de celui-ci. Une modélisation de type CFD, avec suivi lagrangien des cellules n'a de sens que si le modèle du vivant est capable de considérer l'effet de la variation temporelle de lumière sur les microalgues (croissance, métabolisme, photodégradation). L'idée sous-jacente du changement d'échelle, dans ce cas, est de raffiner suffisamment le modèle du vivant, grâce à des expérimentations spécifiques, à très petite échelle afin de les utiliser à des fins prédictives à l'échelle du bioprocédé. Des modèles discrets intermédiaires sont souvent pertinents pour analyser ces données expérimentales. Il faut ensuite valider les prédictions à l'échelle du bioprocédé. De ce fait, nous avons plusieurs briques disponibles non encore publiées. En simulation continue macroscopique, par exemple, deux modèles sont opérationnels (CFD en bioréacteur et Fermentation en Milieu Solide) mais en attente des modèles du vivant pertinents pour être exploités. Parmi les travaux publiés, nous pouvons citer une approche discrète de croissance de champignons(3,4) et la première brique d'un modèle pertinent de croissance de microalgues . Ce dernier sera complété prochainement grâce à des données expérimentales collectées avec un mini-photobioréacteur produit par impression 3D.
Références : (1) Tebbani, S., Lopes, F., Filali, R., Dumur, D., Pareau, D., 2014 - Nonlinear predictive control for maximization of CO2 bio-fixation by microalgae in a photobioreactor. Bioprocess and biosystems engineering, 37, 83-97.
(2) Tebbani, S., Lopes, F., Celis, G. B., 2015 - Nonlinear control of continuous cultures of Porphyridium purpureum in a photobioreactor. Chemical Engineering Science, 123, 207-219.
(3) Du H., Lv P., Ayouz M., Besserer A., Perré P. 2016 - Morphological Characterization and Quantification of the Mycelial Growth of the Brown-Rot Fungus Postia placenta for Modeling Purposes, PLoS ONE, 11, e0162469
(4) Du H., Ayouz M., Lv P., Perré P., 2018 - A lattice-based system for modeling fungal mycelial growth in complex environments, Physica A: Statistical Mechanics and its Applications, 511, 191-206.